Die früheste Erwähnung des 30x-Paradigmas, das ich finden konnte, ist im Original-Sequenzierungspapier für das gesamte Genom von Illumina: Bentley, 2008. In Abbildung 5 zeigen sie insbesondere, dass die meisten SNPs gefunden wurden und dass es bis zum Erreichen des 30-fachen nur wenige nicht abgedeckte / nicht aufgerufene Basen gibt: 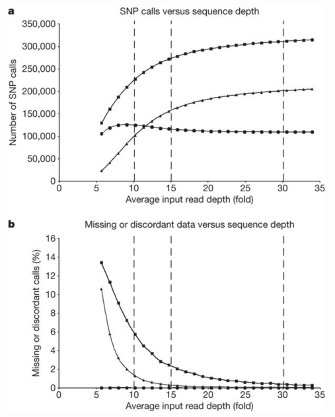
Heutzutage 30x ist immer noch ein gängiger Standard, aber große Keimbahn-Sequenzierungsprojekte drängen oft näher an 25x heran und finden ihn angemessen. Jede Gruppe, die dies ernsthaft tut, hat Leistungsberechnungen basierend auf den Besonderheiten ihrer Maschinen und Vorbereitungen durchgeführt (Dinge wie Fehlerraten und Leselängen sind wichtig!).
Die Krebsgenomik geht in die andere Richtung. Wenn Sie mit Reinheit, Ploidie und subklonalen Populationen zu kämpfen haben, ist viel mehr Abdeckung als 30x erforderlich. Unsere Gruppe hat in dieser Veröffentlichung von 2015 gezeigt, dass selbst bei einer 300-fachen Gesamtgenomabdeckung eines Tumors wahrscheinlich echte seltene Varianten eines Tumors fehlten.
Insgesamt hängt die Sequenzabdeckung, die Sie benötigen, wirklich davon ab, welche Fragen Sie stellen, und ich würde jedem empfehlen, der ein Sequenzierungsexperiment entwirft, sich vorher mit einem Sequenzierungsexperten und einem Statistiker zu beraten (und das ist es auch) noch besser, wenn das die gleiche Person ist!)